 【新智元導讀】清華大學團隊通過AI for Optics創新賦能超分辨光學顯微鏡,將活細胞成像體積分辨率提升15.4倍!藉助這一工具,研究人員可為癌細胞分裂、胚胎髮育等精密生命過程拍攝「4K高清電影」,從而撬動生物醫藥領域的重大突破。
【新智元導讀】清華大學團隊通過AI for Optics創新賦能超分辨光學顯微鏡,將活細胞成像體積分辨率提升15.4倍!藉助這一工具,研究人員可為癌細胞分裂、胚胎髮育等精密生命過程拍攝「4K高清電影」,從而撬動生物醫藥領域的重大突破。
近年來,以深度學習為代表的智能計算方法對光學顯微鏡發展產生變革性影響。通過光學成像系統與智能算法的聯合優化,可在極大程度上突破光學系統設計的時空帶寬固有侷限。
如何基於光學系統與人工智能的交叉創新突破性能瓶頸,以三維視角和亞細胞級各向同性分辨率觀測動態生物過程,是光學顯微成像領域的前沿重要挑戰。
近日,清華大學生命學院李棟課題組與自動化系戴瓊海團隊合作提出一款全新的「AI+」顯微鏡:元學習驅動的反射式晶格光片虛擬結構光照明顯微鏡(Meta-rLLS-VSIM)。合作團隊利用AI賦能超分辨光學顯微鏡,硬件軟件協同優化,實現「1+1>2」的成像效果,開啓了生物醫學活體觀測的全新視角。
論文鏈接:https://www.nature.com/articles/s41592-025-02678-3
在諸多三維光學顯微成像的技術方案中,光片顯微鏡(LSM)是當前最適宜進行多細胞、大體積樣本三維成像的模態之一。
2014年,諾貝爾化學獎得主Eric Betzig在傳統LSM的基礎上發明了晶格光片顯微鏡(LLSM),並通過與結構光照明(SIM)相結合,形成晶格光片結構光照明顯微鏡(LLS-SIM)解決方案。
LLS-SIM可在結構光照明方向上將分辨率提升至150納米左右,這也是當前最新的前沿技術方法。然而,LLS-SIM僅能產生單一方向的結構光照明,導致空間分辨率各向異性,未超分辨方向易產生畸變,限制了對三維亞細胞動態的精準探測。
針對這一難題,Meta-rLLS-VSIM通過AI與光學交叉創新,在不犧牲成像速度、光子代價等核心成像指標的前提下,將LLS-SIM 的一維超分辨能力擴展至XYZ三個維度,實現橫向 120 nm、軸向 160 nm 的近各向同性成像分辨率,相比於LLSM,體積成像分辨率提升15.4倍!
特別地,該工作將元學習策略與系統數據採集過程深度融合,僅需3分鐘就可以完成從訓練數據採集到深度學習模型的自適應部署過程,讓AI工具在實際生物實驗中的應用達到近乎「零門檻」。
合作團隊在植物花粉管、小鼠胚胎、秀麗隱杆線蟲胚胎及真核有絲分裂或分裂間期的其他真核生物等從單細胞到胚胎尺度的多種生物樣本上進行了實驗,驗證了 Meta-LLS-VSIM 在快速、長時程、多色三維超分辨成像任務中的能力。
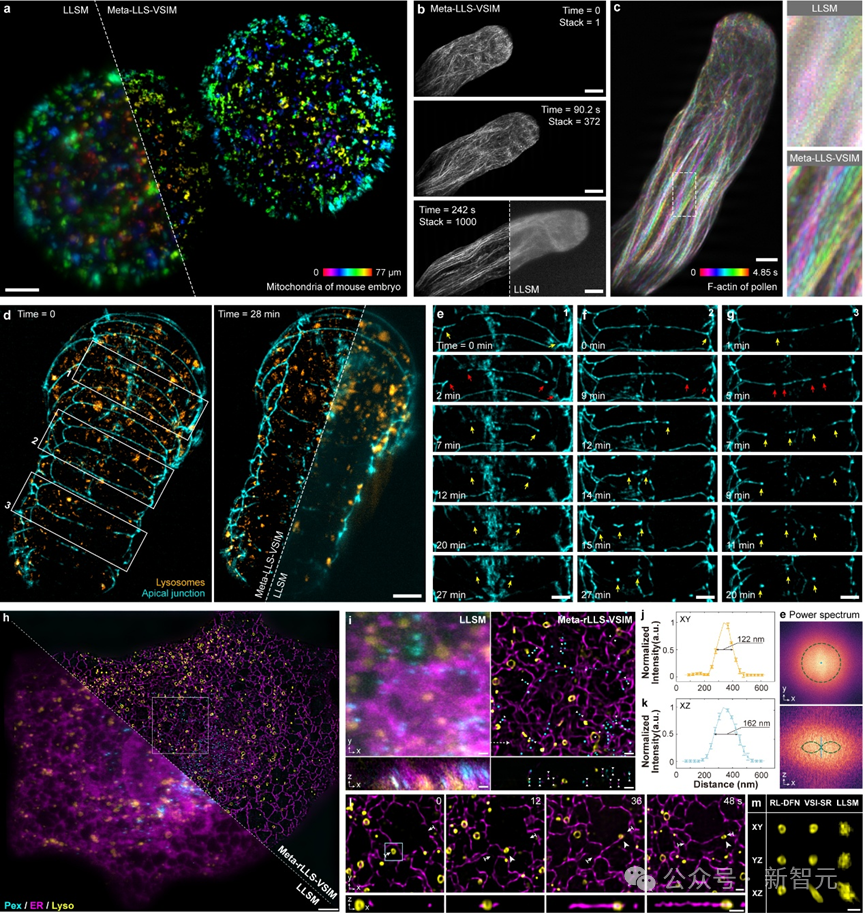
Meta-rLLS-VSIM可實現從細胞到胚胎的跨尺度五維活體超分辨觀測

「虛擬結構光照明」提升橫向分辨率
受限於特殊的雙物鏡垂直安裝的光學系統設計方案,LLS-SIM只能產生固定方向的結構光,而只有結構光照明的方向才能具備超分辨能力。
研究團隊注意到,由於生物樣本生長方向的隨機性,LLS-SIM採集到的單一方向超分辨信息實則涵蓋了各個不同方向上的樣本結構特徵。因而,如果使用深度神經網絡(DNN)可以學習到單一維度超分辨能力,就有潛力將其拓展至其他方向。
於是研究團隊利用自主搭建的成像系統,採集了不同生物樣本的LLSM/LLS-SIM圖像對作為訓練集,並訓練了一個精心設計的DNN模型用以學習單一維度超分辨能力。
在推理階段,只需將LLSM低分辨率圖像以特定取向輸入到DNN模型中,就可以得到這一方向分辨率提升的結果。DNN模型就像是可以任意改變取向的「虛擬結構光」,彌補了LLS-SIM系統的固有缺陷。
最後,通過維納解卷積等方式將不同方向的一維超分辨圖像進行融合,使它們不同方向的超分辨率信息互補,即可獲得在平面內分辨率一致的各向同性超分辨圖像。
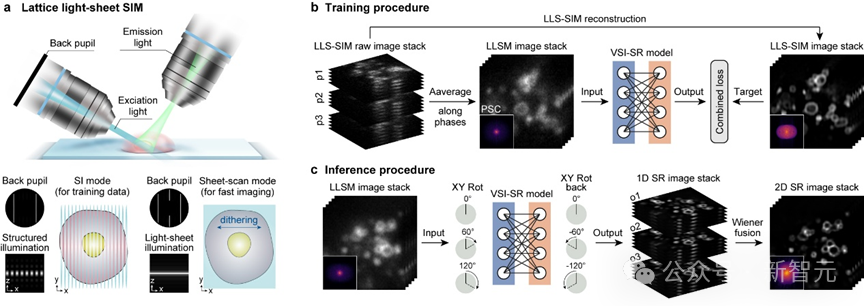
虛擬結構光照明超分辨DNN模型數據採集、訓練與推理
研究團隊在亞細胞結構肌動蛋白微絲上測試了虛擬結構光照明成像(VSI-SR)的效果,發現相比於其它方法,虛擬結構光照明圖像更加清晰,在頻域上不僅包含更多高頻信息,而且保持各向同性。
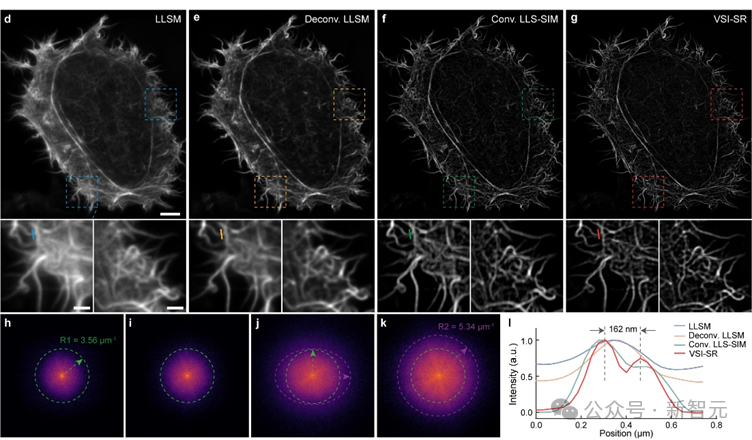
虛擬結構光照明成像效果展示

雙視角信息融合提升軸向分辨率
儘管虛擬結構光照明可將LLS-SIM超分辨能力拓展到整個二維平面,但無法提升與這一平面垂直方向上的分辨率(即軸向分辨率)。
為解決這一問題,研究團隊將承載生物樣本的蓋玻片替換為一個反射鏡,在進行三維成像時,鏡面上方的實像和下方的虛像被同時拍攝,並且二者的空間視角不同。
雙視角之於單視角類似於雙目視覺之於單目視覺,兩個視角的分辨率空間取向不同,意味着兩個視角橫向與軸向分辨率互補,融合兩個視角圖像的信息可在空間信息採集的本質上提升軸向分辨率。

Meta-rLLS-VSIM基於鏡面反射的雙視角成像原理
為了更好的融合兩個視角信息,研究團隊提出了光學成像模型啓發的Richardson-Lucy雙循環融合網絡架構(RL-DFN)。
RL-DFN融合了多視角Richardson-Lucy迭代公式,使用可訓練卷積層替代前向和反向傳播算子,在保證信息融合符合統計學規律的前提下,提升融合效果。
隨後,將融合得到的高分辨率結果與光學系統的點擴散函數(PSF)進行卷積,並與輸入圖像體棧計算循環損失(Cycle Loss)。這一Cycle Loss將RL-DFN約束為滿足物理先驗條件的解卷積模型,使軸向分辨率的提升在物理上可靠。
為進一步提升融合結果保真度,研究團隊在RL-DFN設計中引入了判別器。判別器通過分辨RL-DFN輸出的任意二維切面圖與虛擬結構光成像得到的各向同性超分辨平面圖,與RL-DFN相互競爭。
而RL-DFN為了騙過判別器,會努力提升信息融合效果,使任意方向的切面都達到各向同性超分辨,最後實現在三維空間上的各向同性分辨率。
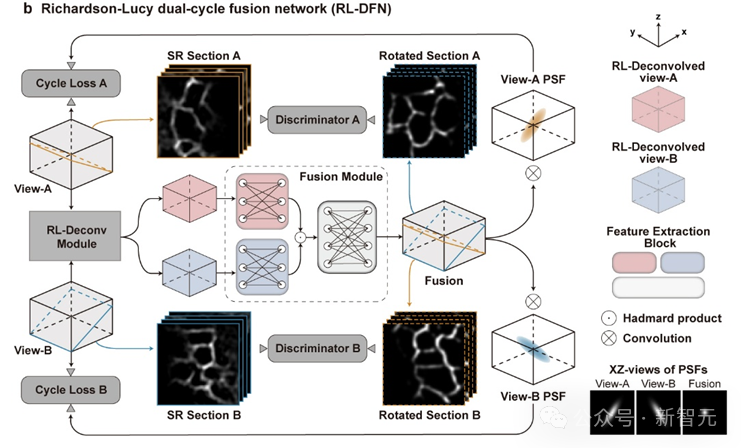
RL-DFN的模型結構示意
在雙視角循環Cycle Loss的約束和判別器的對抗下,RL-DFN可以充分利用雙視角互補分辨率信息,合理提升軸向分辨率。
研究團隊將RL-DFN與其他無監督方法在仿真數據上進行對比,發現RL-DFN能夠實現各向同性的超分辨信息融合,並且更精確地恢復出樣本細節。
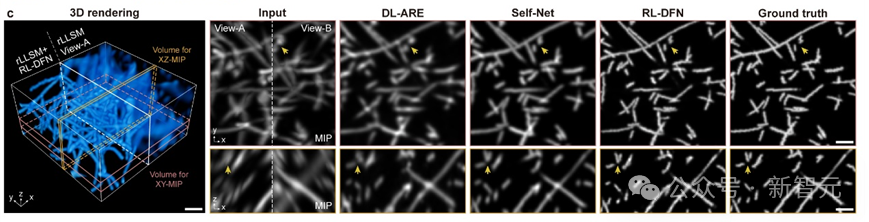
RL-DFN與其它方法重建效果對比

元學習實現AI模型的快速自適應部署
在實際生物成像實驗當中,由於DNN泛化性和表徵能力受限,通常需要為每一種不同的生物結構、甚至不同信噪比訓練專用模型,以得到最佳的模型推理效果。
而亞細胞結構成百上千,且形態各異,為每一種結構訓練模型需採集海量訓練數據,並消耗大量計算成本和時間成本,導致AI工具在實際成像實驗中難以被廣泛應用。
為了解決這一難題,研究團隊將「元學習」與光學顯微系統的數據採集時序深度融合,將模型訓練所需的數據量降低12倍,訓練時間降低720倍,實現了AI模型的快速自適應部署。
元學習的核心思想是在訓練過程中不僅關注單一任務的性能優化,而是希望使訓練模型在面對新任務時,快速地調整自身參數,以實現模型的快速遷移。
元學習訓練得到的通用元模型雖然無法在單個任務上達到最佳的推理效果,但是它可以通過在少量子任務數據的少量訓練,快速收斂到子任務的最佳模型。通用元模型相當於「交通樞紐」,有着到達所有子任務最佳模型的「快速路徑」。
在Meta-rLLS-VSIM中,研究團隊將不同生物樣本和信噪比的超分辨重建問題視為單獨的子任務,訓練單方向超分辨任務的通用元模型。
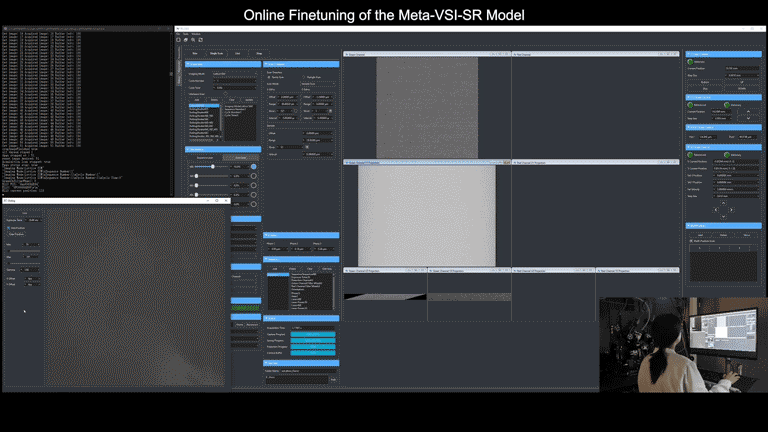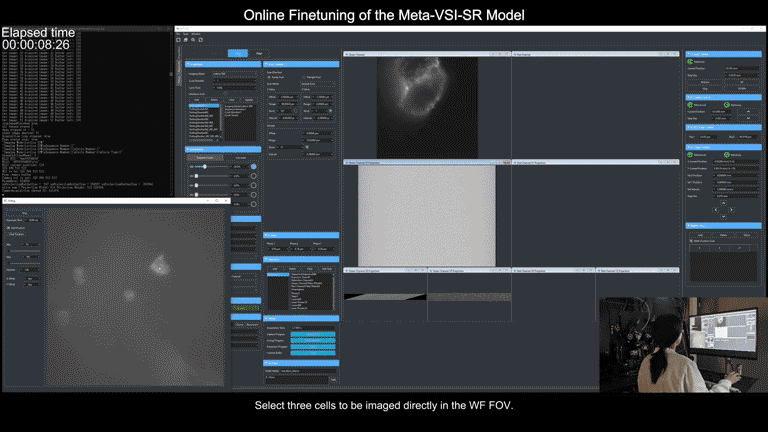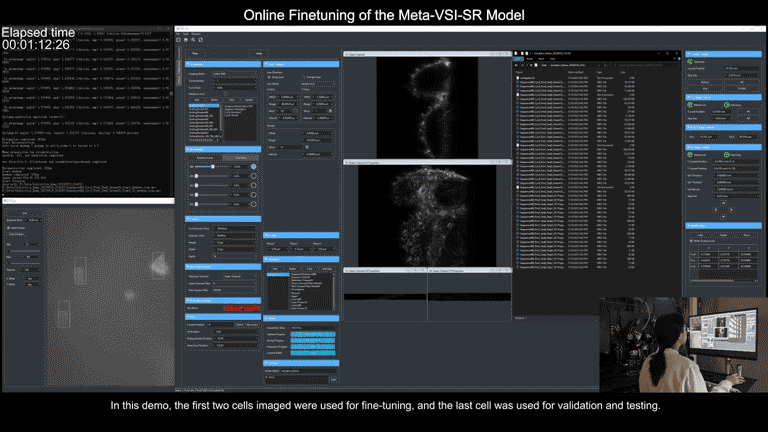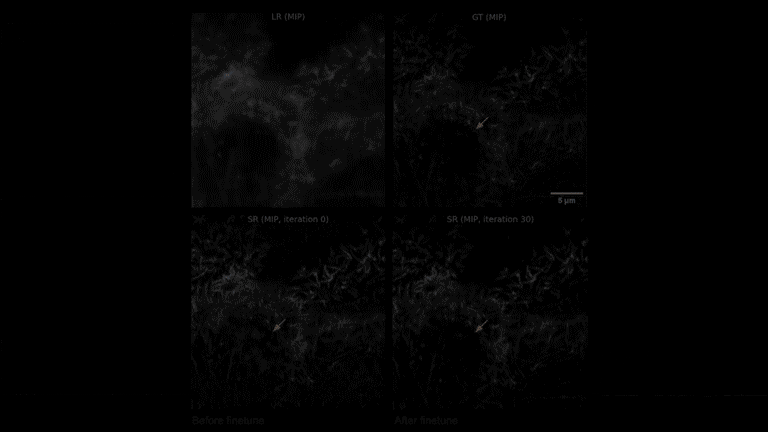
當在新的子任務部署時,僅需採集3對高低分辨率圖像對,並進行30 次微調迭代,就可以快速得到針對這一新任務的最優DNN模型。
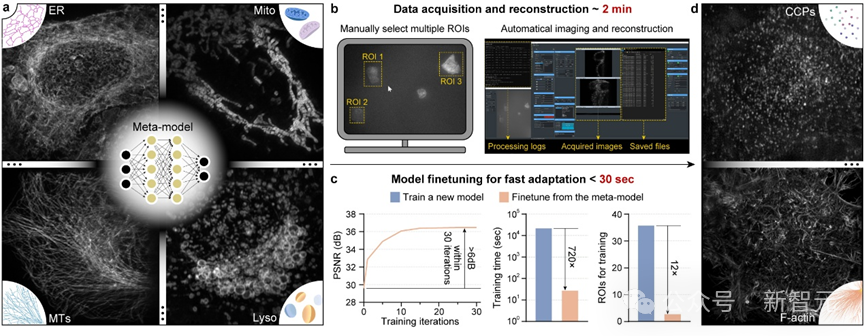
元學習驅動的模型快速自適應部署實現流程
為了簡化操作流程,研究團隊還在顯微鏡控制程序上實現了通用元模型部署流程的全自動化。使用者只需要在圖形界面點擊幾個待採集區域,後續從數據採集、到訓練集超分辨重建、再到元模型微調,全部流程都可在後端自動實現,且僅需不到3分鐘時間。
元學習驅動的模型快速自適應部署過程視頻展示
藉助元學習構建的「交通樞紐」和全自動化部署流程,研究團隊實現了AI模型在不同生物樣本場景下的快速自適應部署,大大降低了AI模型在實際生物成像實驗中的使用門檻,讓AI真正可以輔助生命科學新發現。
綜上,Meta-rLLS-VSIM通過反射增強雙視角晶格光片顯微鏡與元學習快速自適應部署模式的成像系統硬件升級,與虛擬結構光照明和RL雙循環融合網絡的人工智能算法創新,展現了軟硬件協同優化帶來的光學顯微鏡成像能力巨大提升。
Meta-rLLS-VSIM的出現為細胞生物學、神經科學等基礎學科發展提供了新的技術路徑,有望幫助生命科學研究人員從更全面的多維視角發現、理解、探究豐富多彩的生物現象。
參考資料:
https://www.nature.com/articles/s41592-025-02678-